Solo en 2024, se publicaron más de 500 nuevos genomas vegetales. En medio de ese ritmo vertiginoso, investigadores europeos lanzaron PubPlant, una herramienta gratuita y dinámica, con actualización permanente, que permite explorar, analizar y comprender el mapa genético de las plantas como nunca antes.
ChileBio / 3 de julio, 2025.- En el mundo de la ciencia vegetal, las últimas dos décadas han sido una auténtica revolución. Desde que se publicó por primera vez el genoma de Arabidopsis thaliana en el año 2000, más de mil especies de plantas han sido secuenciadas, muchas de ellas gracias a tecnologías que hace pocos años eran impensables. Sin embargo, este avance ha traído consigo un nuevo desafío: ¿cómo organizar tanta información, mantenerla al día y hacerla accesible para todos?
La respuesta llegó este año desde Europa, donde un grupo de investigadores presentó PubPlant, una base de datos en línea que busca convertirse en el punto de encuentro para toda la información genómica vegetal publicada hasta la fecha. La plataforma no solo reúne los genomas ya secuenciados, sino que también los organiza, los pone en contexto evolutivo y los actualiza mensualmente.
[Recomendado: Investigadores chinos lanzan «PlantGPT», un avanzado asistente de IA especializado en genómica funcional de plantas]
Publicado en la revista Frontiers in Plant Science, el proyecto fue liderado por Markus Schwacke, Thomas Usadel y otros científicos especializados en bioinformática y genómica vegetal. El objetivo es simple pero ambicioso: que cualquier persona interesada (ya sea investigador, estudiante o profesional del agro) pueda navegar por el universo genético de las plantas de forma intuitiva y confiable.
Un mapa que se actualiza mensualmente
Lo más innovador de PubPlant es su capacidad para mantenerse al día. Mientras que las revisiones científicas tradicionales suelen quedar desactualizadas a los pocos meses de publicarse, esta herramienta se actualiza mensualmente con las últimas publicaciones sobre secuenciación de genomas vegetales completos.
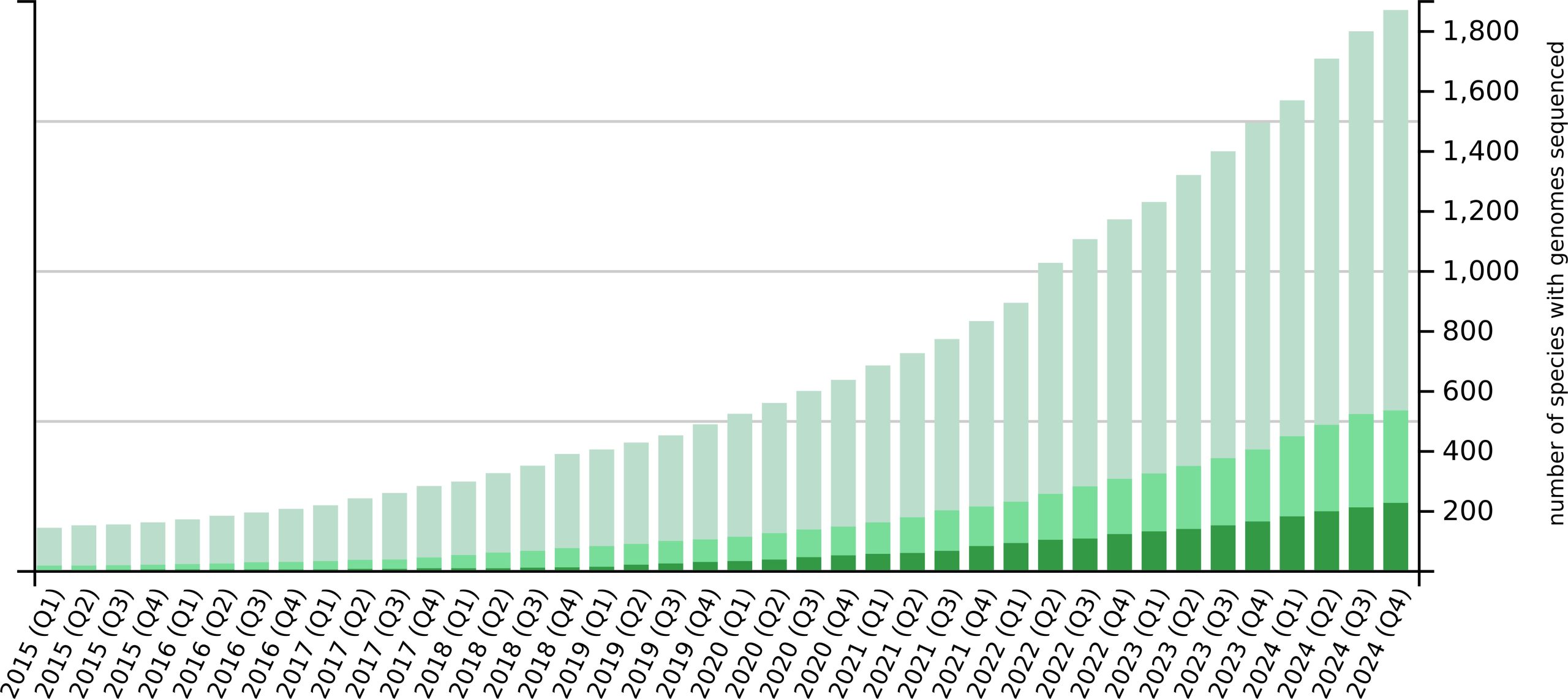
El estudio destaca que, tras la publicación del genoma de Arabidopsis thaliana en el año 2000, tardamos una década en alcanzar apenas 20 genomas secuenciados. Sin embargo, solo hicieron falta otros cuatro años para superar los 100, y para el año 2020 ya se había alcanzado el hito de 500 especies. Desde entonces, el crecimiento ha sido exponencial: en los dos años siguientes se sumaron otros 500 genomas, un logro que antes había tomado veinte años.
Para ilustrar la magnitud del avance, basta mirar los datos más recientes: solo en 2024 se publicaron más de 500 nuevos genomas, de los cuales 370 correspondían a especies secuenciadas por primera vez. A ese ritmo, contar con una base de datos viva y actualizada como PubPlant no es solo útil, sino que se vuelve esencial para la investigación agrícola.
Agricultura y biodiversidad bajo la lupa
Al explorar la base, uno de los primeros patrones que salta a la vista es el fuerte sesgo hacia especies cultivadas. Familias como las gramíneas (trigo, arroz, maíz), las leguminosas (soja, poroto), las solanáceas (papa, tomate) o las brasicáceas (coliflor, rábano) están ampliamente representadas. Y tiene sentido: son las especies que más alimentan al planeta.
Pero al mismo tiempo, PubPlant deja en evidencia que muchas otras plantas (desde árboles nativos hasta hierbas medicinales) siguen esperando su turno para ser estudiadas en profundidad. Esta herramienta permite visualizar esas lagunas de conocimiento, lo que puede orientar futuras investigaciones, sobre todo en un contexto de crisis climática y búsqueda de nuevas fuentes alimenticias.
Fácil de usar, incluso para quienes no son expertos
Una de las claves del proyecto fue su enfoque en la accesibilidad. PubPlant fue diseñado para que cualquier persona pueda navegarlo sin necesidad de tener conocimientos técnicos avanzados. Con solo un par de clics se pueden explorar líneas de tiempo interactivas, árboles evolutivos, mapas de distribución taxonómica y enlaces directos a las publicaciones científicas originales.
Cada entrada incluye información detallada: nombre científico, fecha de publicación, tamaño del genoma, método de secuenciación y enlaces a bases de datos externas como Ensembl Plants o NCBI.
Una brújula para la investigación vegetal
Los autores del artículo subrayan que PubPlant es un recurso abierto, gratuito y comunitario. Su éxito dependerá de la participación activa de la comunidad científica: compartiendo nuevas publicaciones, proponiendo mejoras y utilizando sus herramientas para diseñar estrategias de investigación vegetal más equilibradas y sostenibles.
En un mundo donde la agricultura enfrenta retos sin precedentes (sequías, degradación de suelos, nuevas plagas) conocer el genoma de las plantas no es solo una curiosidad académica, sinouna necesidad estratégica. PubPlant llega para ser la brújula abierta en esa jungla creciente de datos genómicos.

