Durante los casi 20 años transcurridos desde la comercialización del primer producto obtenido de una planta transgénica, la biología, y muy particularmente la genómica vegetal, ha experimentado un avance espectacular. La secuenciación de los genomas de distintas plantas y la resecuenciación de un buen número de variedades de cada una de ellas está permitiendo obtener una información muy valiosa sobre los alelos de genes que se han seleccionado a lo largo de la historia y que explican algunas de las características de las plantas cultivadas actuales. Pero por otro lado, el avance científico ha permitido desarrollar tecnologías que pueden tener aplicaciones importantes en la mejora genética de plantas.
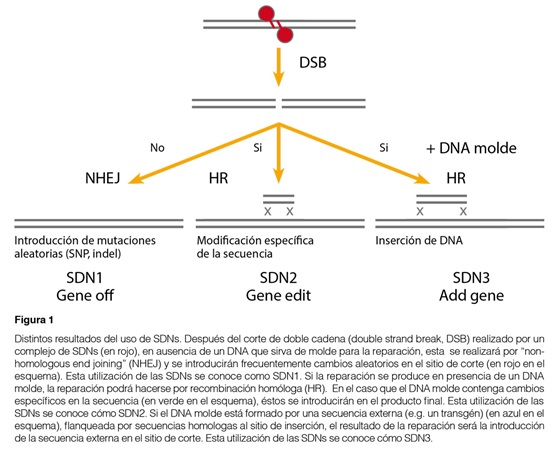
Entre las nuevas técnicas de mejoramiento genético vegetal basadas en biotecnología destacan por su potencial las técnicas de mutagénesis y, en particular, las técnicas de edición de genomas basadas en nucleasas específicas o SDNs (Site Directed Nucleases). Las SDNs son nucleasas capaces de cortar la doble cadena del ADN de manera específica en un sitio predefinido del genoma gracias a su capacidad de reconocer secuencias específicas. La reparación del sitio de inserción por los mecanismos celulares resulta frecuentemente en la incorporación de mutaciones en el sitio de corte. De esta manera se pueden obtener mutaciones en sitios específicos del genoma. En presencia de un ADN homólogo que pueda servir de molde, las roturas del ADN se reparan frecuentemente por recombinación homóloga (HR). Por ello si además de la SDN se suministra un ADN molde que incorpore cambios específicos, se incorporarán estos cambios en el sitio de corte y se habrá podido introducir mutaciones bien definidas en sitios específicos del genoma. De la misma forma, se pueden introducir secuencias largas de ADN, por ejemplo un transgén, en el sitio de corte de la SDN si se añaden a ambos lados de la secuencia de interés, secuencias homólogas al sitio de corte. Estas aplicaciones han venido en llamarse SDN1 (mutación aleatoria en un sitio predefinido), SDN2 (mutación específica en un sitio predefinido) y SDN3 (incorporación de una secuencia en un sitio predefinido) (ver Figura 1).
Las primeras SDNs que se utilizaron fueron las meganucleasas, un tipo particular de nucleasas naturales codificadas por ciertos intrones móviles que poseen un sitio de reconocimiento de 20-30 bp. Sin embargo, la poca flexibilidad de estas proteínas y la dificultad de modificarlas para reconocer sitios distintos a los naturales ha limitado su aplicación. Esta limitación ha quedado superada sucesivamente por nuevos tipos de SDNs, en primer lugar por las Zinc-Finger Nucleases (ZFNs), que son proteínas quiméricas compuestas por un dominio de unión al DNA de tipo dedo de zinc y una nucleasa convencional (frecuentemente FokI). Los dominios dedos de zinc interaccionan específicamente con un triplete de bases del DNA y su especificidad es conocida, por lo que se pueden diseñar multímeros de dedos de zinc que reconozcan secuencias específicas. Pero la plasticidad de las ZFNs, aun siendo mucho mayor que la de las meganucleasas, es relativamente limitada y la obtención de ZFNs específicas es costosa, por lo que estas proteínas se vieron rápidamente superadas por un nuevo tipo de SDN, las TALENs. Las transcription-activatorlike (TAL) effector nucleases son proteínas quiméricas entre un dominio nucleasa (de nuevo, frecuentemente FokI) y un dominio de unión al DNA derivado de los efectores TAL de Xantomonas, unos factores de transcripción que permiten a la bacteria reprogramar específicamente la transcripción de las células de una planta infectada. Estas proteínas reconocen el DNA mediante unos dominios que siguen un código simple entre tres residuos proteicos específicos para cada nucleótido. Es por lo tanto relativamente sencillo construir multímeros de los dominios proteicos para reconocer virtualmente cualquier secuencia. Sin embargo todas estas SDNs se han visto superadas rápidamente por las CRISPR/Cas. Se trata en este caso de un proceso usado por algunas bacterias para eliminar virus o plásmidos invasivos. El dominio de unión al DNA que dirige a la nucleasa no es en este caso un dominio proteico sino que es un RNA (RNA guía). Esto convierte al sistema CRISPR/ Cas en un método altamente flexible y fácil de adaptar con el que se puede cortar específicamente cualquier secuencia sintetizando un RNA guía complementario. El enorme potencial de las técnicas de edición genómica basadas en el uso de CRISPR/Cas llevó a la revista Science a elegirla como el principal avance del año 2015.
En el caso de la mejora genética de plantas, la gran cantidad de nuevos genotipos de plantas de interés agronómico publicados en revistas científicas en los últimos años muestra el enorme impacto que esta tecnología puede tener en un futuro próximo en el desarrollo de plantas comerciales. Pero además de permitir la obtención de mutantes de una manera dirigida y permitir generar de manera específica nuevos alelos de genes que son responsables de fenotipos conocidos, estas técnicas se pueden usar también para explorar nuevos alelos o nuevas combinaciones de alelos no conocidos. Sin embargo, para que puedan llegar al campo es necesario que estas nuevas metodologías encuentren un marco regulatorio apropiado.
La edición de genomas puede dar lugar a plantas que sean indistinguibles de aquellas que contiene variantes génicas aparecidas de forma espontánea. No parece razonable que los requerimientos exigidos sean los mismos que para las modificaciones que se realizan con construcciones génicas que incluyen nuevas secuencias de origen a veces lejano de la especie transformada.
Referencias
van de Wiel CCM, Schaart JG, Lotz LAP, Smulders MJM. 2017. New traits in crops produced by genome editing techniques based on deletions. Plant Biotechnol Rep. 11(1):1-8.
Mohanta TK, Bashir T, Hashem A, Abd Allah EF, Bae H. 2017. Genome Editing Tools in Plants. Genes (Basel). 8(12). pii: E399.
Zhang K, Raboanatahiry N, Zhu B, Li M. 2017. Progress in Genome Editing Technology and Its Application in Plants. Front Plant Sci. 8:177.
Podevin N, Davies HV, Hartung F, Nogué F, Casacuberta JM. 2013 Site-directed nucleases: a paradigm shift in predictable, knowledgebased plant breeding. Trends Biotechnol. 31: 375–83.