Tener más de dos juegos de cromosomas puede ayudar a las plantas a adaptarse y evolucionar, pero generar nuevos cultivos con este tipo de genoma es un desafío. Se ha desarrollado una hoja de ruta para hacer precisamente eso aplicando edición genética en variedades de arroz silvestre.
Nature / 5 de marzo, 2021.- A veces, todos deseamos poder hacer más de una cosa a la vez: hacer mandados, ponernos al día con los plazos del trabajo y tal vez tomarnos ese café con un amigo que tanto tiempo hemos esperado. Un estado genético conocido como poliploidía ayuda a algunos genomas de plantas a hacer precisamente esto. La mayoría de las plantas, como los humanos, son diploides, es decir, cuentan con dos juegos de cada cromosoma. Pero las plantas poliploides tienen cuatro, seis o incluso ocho juegos de cromosomas.
Estas adiciones permiten que diferentes copias de un gen asuman diferentes roles y proporcionan un amortiguador contra mutaciones potencialmente dañinas. En consecuencia, la poliploidía ha servido como un modo común de evolución en las plantas con flores [1]. Yu et al.[2], publicado en Cell, describe un enfoque viable para producir una forma domesticada de arroz poliploide mediante la edición de genes. Su avance podría permitirnos cosechar los beneficios de la poliploidía en una de las especies de cultivos más importantes del mundo.
[Recomendado: CRISPR permite la domesticación de plantas silvestres en poco tiempo]Todas las especies de cultivos evolucionaron a partir de ancestros silvestres, a medida que los humanos salvaron y propagaron plantas que tenían atributos favorables (pérdida de los mecanismos de dispersión de semillas, por ejemplo, y semillas y frutos más grandes[3]) durante cientos o miles de años. El principal cultivo de arroz del mundo, la especie asiática Oryza sativa, fue domesticado hace unos 9.000 años a partir de su progenitor silvestre, Oryza rufipogon, a través de procesos que se cree que se han producido en varias regiones de Asia [4,5]. Ambas especies son diploides y llevan dos juegos de 12 cromosomas.
Para los científicos del arroz, la idea de desarrollar arroz cultivado poliploide es tentadora como un medio potencial para la mejora de cultivos en el futuro, especialmente frente a la variabilidad climática [6]. Las copias de genes adicionales de la planta podrían permitir una rápida adaptación en respuesta a cambios importantes en el medio ambiente sin la pérdida de características favorables [7]. Pero generar un arroz poliploide a partir de una planta diploide cultivada es un gran desafío técnico. Con eso en mente, Yu et al. adoptó un enfoque completamente diferente. Los autores comenzaron con un primo poliploide silvestre lejano de O. sativa y O. rufipogon, y lo domesticaron utilizando enfoques biotecnológicos (Fig. 1).
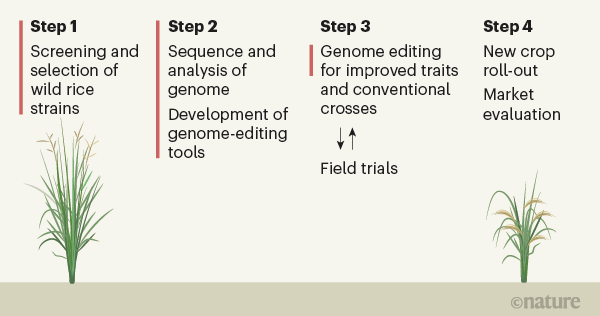
Los autores primero dedicaron tiempo a identificar una cepa inicial adecuada. El candidato ideal debía ser susceptible de inducción y regeneración de callos, un proceso en el que se cultivan tejidos vegetales para producir una masa de células parcialmente indiferenciadas llamada callo, a partir de la cual se generan nuevas plantas. Estas propiedades son esenciales para las técnicas de edición de genes. El individuo seleccionado también necesitaba tener una alta biomasa y tolerancia a diversos estreses abióticos y bióticos, por ejemplo, resistencia al calor y a los insectos. Después de seleccionar 28 líneas de arroz silvestre poliploide, se seleccionó una cepa de Oryza alta y se denominó arroz poliploide 1 (PPR1).
[Recomendado: Desarrollan un nuevo cultivo a partir de un tomate silvestre con edición genética]Oryza alta tiene cuatro juegos de cromosomas (es tetraploide) y se encuentra en Centro y Sudamérica [8]. La especie surgió como resultado de la hibridación entre dos ancestros que tenían genomas diploides, denominados C y D. La cepa PPR1 seleccionada por Yu et al. se ve bastante diferente de la cultivada O. sativa. Por ejemplo, es muy alto: más de 2,7 metros, en comparación con 1 metro o menos de la típica O. sativa. Produce abundante biomasa y tiene hojas anchas y pequeñas semillas escasas adornadas con aristas (protuberancias puntiagudas que se cree que ayudan a la diseminación de las semillas). Como tal, domesticar a este pariente salvaje no fue poca cosa.
Yu y sus colegas establecieron métodos para la edición de genes en PPR1 y ensamblaron un genoma de alta calidad para la cepa. Esto actuó como un mapa que ayudó a identificar genes a los que apuntar para la domesticación. Los autores compararon PPR1 con un genoma de O. sativa denominado Nipponbare. Descubrieron alrededor de 10,000 genes en cada uno de los genomas C y D que no tenían equivalentes (homólogos) en Nipponbare. Por el contrario, alrededor de 39.500 genes en Nipponbare (70,41% del genoma) tenían homólogos en PPR1.
Este último fue un resultado prometedor, porque significaba que los genes responsables de la domesticación en O. sativa probablemente tenían versiones relacionadas en PPR1. Los investigadores editaron un conjunto de genes de este tipo en PPR1 que se sabía que estaban involucrados en la domesticación de O. sativa. Esto condujo a una serie de mejoras en PPR1: pérdida de rotura (un mecanismo de dispersión de semillas), de modo que las semillas no se caen de la planta antes de la cosecha; longitud de la arista reducida para facilitar el procesamiento poscosecha; mayor longitud de grano para granos más grandes y mayor rendimiento; disminución de la altura y aumento del diámetro del tallo para soportar los granos más pesados; y tiempos de floración modificados (tanto más largos como más cortos), necesarios para la adaptación local a diferentes latitudes.
[Recomendado: Avanzan en desarrollo de «arroz C4»: genéticamente modificado con una ruta fotosintética más eficiente]Juntos, los esfuerzos de Yu y sus colegas llevaron a la producción de líneas PPR1 con características domesticadas en solo unas pocas generaciones, acelerando un proceso que generalmente ocurre durante cientos o miles de años. El trabajo abre la puerta al desarrollo de plantas que no solo pueden resistir mejor las tensiones ambientales (una característica crucial para la seguridad alimentaria mundial frente a los climas cambiantes), sino que también podrían tener otras características (por ejemplo, una mejor nutrición y sabor) que podrían ayudar arroz para satisfacer las preferencias cambiantes de los consumidores en el futuro. Además, la estrategia que los autores han ideado podría teóricamente proporcionar una hoja de ruta para aplicar la biotecnología para impulsar la domesticación de parientes silvestres de otros cultivos actuales.
Las técnicas establecidas por Yu et al. aguardan pruebas en otras cepas de arroz tetraploide silverstre. Será necesaria una extensión exitosa a un acervo genético más amplio para que los investigadores y los mejoradores generen un repositorio diverso de poliploides domesticados, que luego podrían usarse para generar cepas mejoradas mediante cruces convencionales o edición del genoma: cepas adaptadas a sistemas de producción particulares, por ejemplo, o aquellos con alta aceptabilidad en el mercado.
[Recomendado: Arroz genéticamente modificado resistente al calor también produce un 20% más de grano]Y aunque los poliploides silvestres son muy prometedores como fuentes de genes aún sin explotar que confieren tolerancia a estreses abióticos como la sequía, es probable que estos rasgos sean complejos, como señalan los autores, y que estén influenciados por muchos genes, cada uno de los cuales tiene solo un pequeño efecto. Se necesita un conocimiento más profundo de la genética de estas plantas para poder apreciar todo el potencial de los arroces silvestres.
Hay un largo camino por delante para la mejora de arroz poliploide domesticado. Pero ya se han sembrado las primeras semillas. A medida que aumenta la demanda de sistemas alimentarios ágiles y resilientes, la domesticación rápida y la mejora de las especies de plantas silvestres, incluidos los poliploides, pueden convertirse en un valioso instrumento en la caja de herramientas de la agricultura.
Referencias:
Wood, T. E. et al. Proc. Natl Acad. Sci. USA 106, 13875–13879 (2009).
Yu, H. et al. Cell 184, 1156–1170 (2021).
Doebley, J. F., Gaut, B. S. & Smith, B. D. Cell 127, 1309–1321 (2006).
Londo, J. P., Chiang, Y. C., Hung, K. H., Chiang, T. Y. & Schaal, B. A. Proc. Natl Acad. Sci. USA 103, 9578–9583 (2006).
- Huang, X. et al. Nature 490, 497–501 (2012).
Koide, Y., Kuniyoshi, D. & Kishima, Y. Front. Plant Sci. 11, 1231 (2020).
Van de Peer, Y., Mizrachi, E. & Marchal, K. Nature Rev. Genet. 18, 411 (2017).
Vaughan, D. A. The Wild Relatives of Rice: A Genetic Resources Handbook (Int. Rice Res. Inst., 1994).


